KAIST
BREAKTHROUGHS
Research Webzine of the KAIST College of Engineering since 2014
Fall 2024 Vol. 23암은 정상세포와 다르게 세포 내 비정상적인 대사 반응들이 일어나며, 이에 대한 연구가 진단과 치료를 목적으로 활발히 진행되고 있다. 본 연구에서는 컴퓨터 대사모델을 이용하여, 암 체세포 유전자 돌연변이와 관련된 새로운 대사물질 및 대사경로를 예측하는 컴퓨터 방법론을 개발하였다.
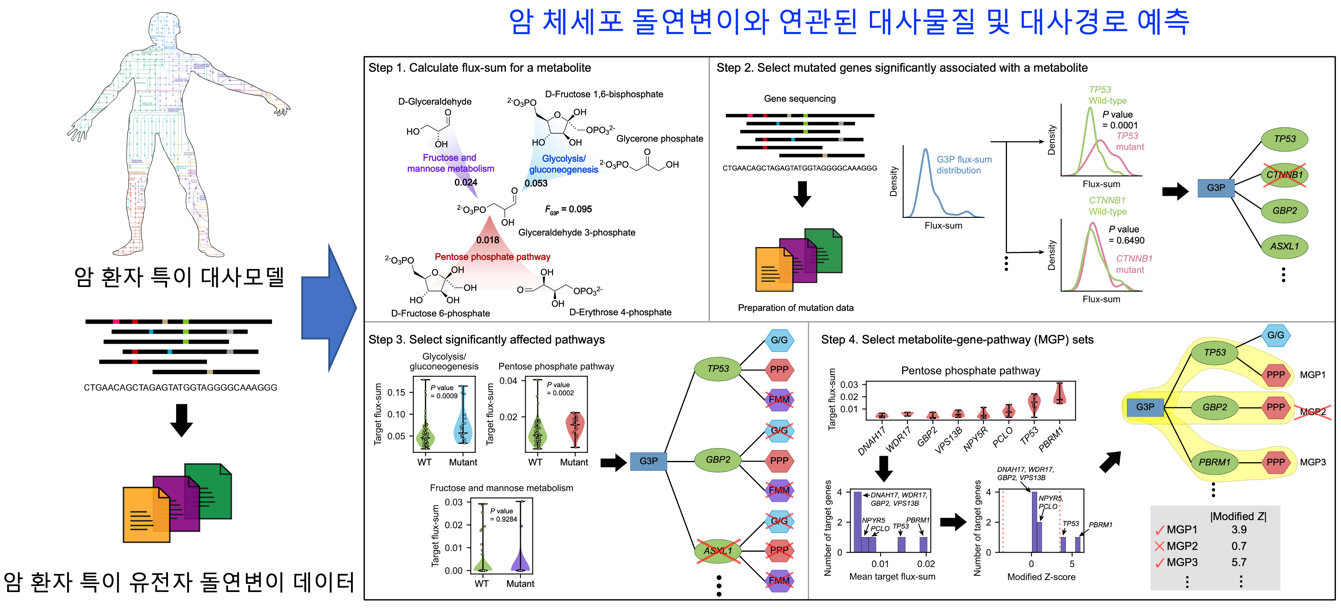
암은 정상세포와 다르게 세포 내 비정상적인 대사 반응들이 일어나며, 이에 대한 연구가 진단과 치료를 목적으로 활발히 진행되고 있다. 우리 대학 생명화학공학과 김현욱 교수, 이상엽 특훈 교수 연구팀은 서울대학교병원 고영일 교수, 윤홍석 교수, 정창욱 교수 연구팀과 협력하여 24개 암종에 해당하는 1,043명의 암 환자 대사모델을 구축했다. 대사모델은 세포 내 모든 대사 반응을 예측할 수 있는 컴퓨터 모델이다. 본 공동 연구팀은 대사모델을 이용하여, 암 체세포 유전자 돌연변이와 관련된 새로운 대사물질 및 대사경로를 예측하는 컴퓨터 방법론을 개발하였다.
최근 FDA 승인을 받은 '팁소보(ivosidenib)'와 '아이드하이파(enasidenib)' 같은 항암제는 암 관련 대사물질(oncometabolite)을 표적으로 한다. 이러한 대사물질은 유전자 돌연변이로 인해 비정상적으로 축적되며, 암세포의 성장과 생존을 촉진한다. 대표적인 암 관련 대사물질로는 2-하이드록시글루타레이트(2-hydroxyglutarate), 숙시네이트(succinate), 푸마레이트(fumarate) 등이 있다.
암 대사 연구와 암 관련 대사물질 발굴에는 많은 시간과 비용이 소요되기 때문에, 현재까지 밝혀진 암 관련 대사물질은 극소수에 불과하다. 본 공동 연구팀은 국제 암 연구 컨소시엄의 데이터를 통합해 게놈 수준의 대사모델을 만들었다. 이후 1,043명의 암 환자 특이 대사모델과 해당 환자들의 돌연변이 데이터를 활용해 4단계의 컴퓨터 방법론을 개발했다. 첫째, 암 환자 대사모델을 시뮬레이션하여 각 대사물질의 활성을 예측한다. 둘째, 유전자 돌연변이가 대사물질 활성에 미치는 유의한 영향을 분석한다. 셋째, 유전자 돌연변이와 연관된 대사경로를 선별한다. 마지막으로, '유전자-대사물질-대사경로' 조합을 완성한다.
이 방법론은 다른 암종에도 쉽게 적용할 수 있으며, 유전자 돌연변이가 대사경로를 통해 세포 대사에 미치는 영향을 체계적으로 예측할 수 있는 최초의 방법론이다. 연구 결과는 향후 암 대사 및 암 유발 대사물질 연구의 중요한 참고 자료가 될 것이다.